# Análisis de Mediación {#sec-mediation}
## Objetivos de aprendizaje
Al finalizar este capítulo, serás capaz de:
- Distinguir entre efectos directos e indirectos
- Definir efectos de mediación causal (NDE, NIE)
- Identificar las asunciones para mediación
- Aplicar el paquete `mediation` en R
## ¿Qué es la mediación?
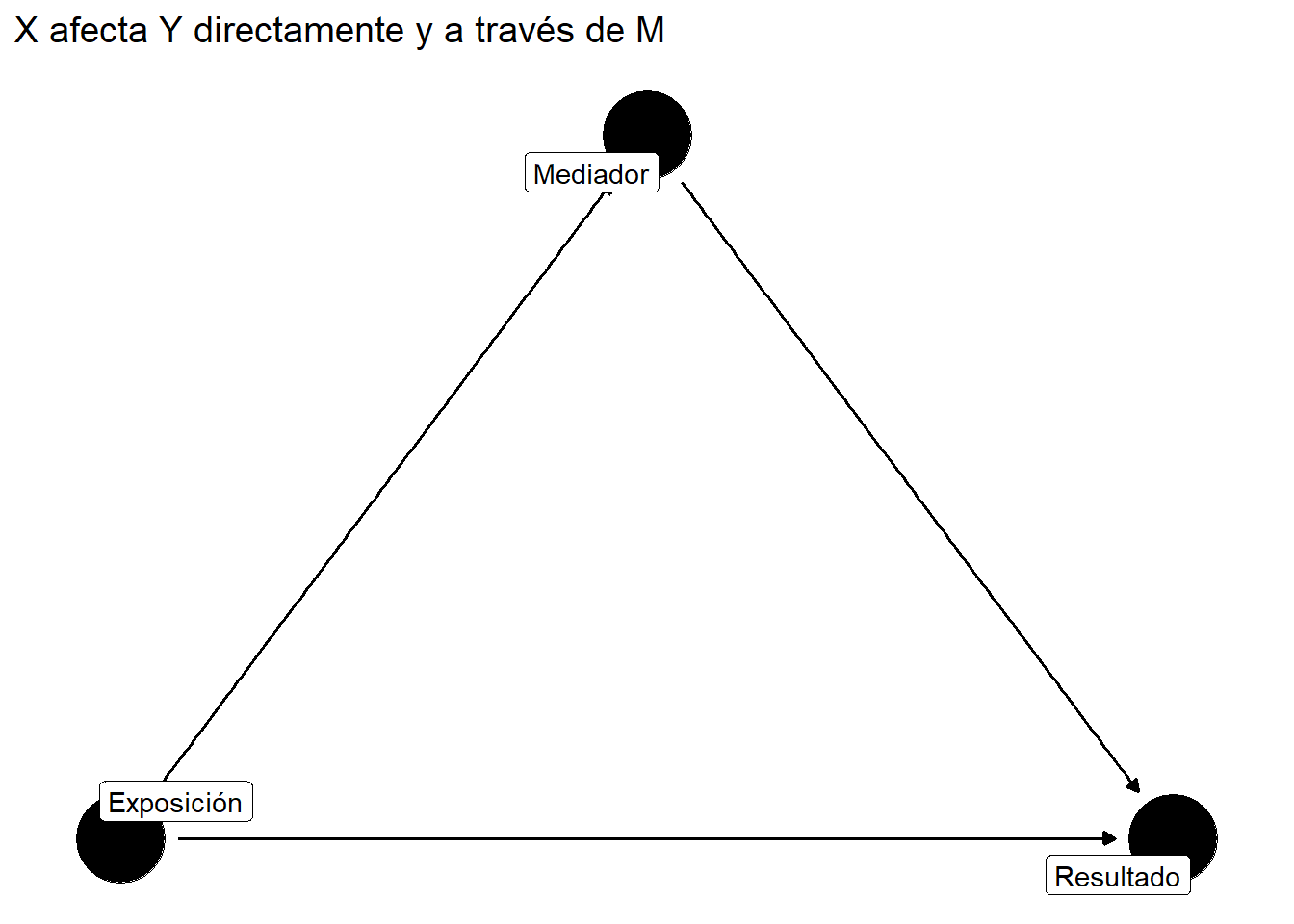
La **mediación** examina *cómo* o *por qué* una exposición afecta un resultado. El efecto puede ser:
- **Directo**: X → Y
- **Indirecto**: X → M → Y (a través de un mediador)
```{r}
#| label: fig-mediation-dag
#| fig-cap: "Diagrama de mediación simple"
#| code-fold: true
library(ggdag)
library(ggplot2)
med_dag <- dagify(
M ~ X,
Y ~ X + M,
coords = list(
x = c(X = 0, M = 1, Y = 2),
y = c(X = 0, M = 0.5, Y = 0)
),
labels = c(
X = "Exposición",
M = "Mediador",
Y = "Resultado"
)
)
ggdag(med_dag, text = FALSE, use_labels = "label") +
theme_dag() +
labs(title = "X afecta Y directamente y a través de M")
```
## Definiciones causales
### Efectos naturales (NDE y NIE)
- **Efecto Directo Natural (NDE)**: Efecto de X sobre Y manteniendo M en el nivel que habría tenido sin la exposición
- **Efecto Indirecto Natural (NIE)**: Efecto de cambiar M desde el nivel sin exposición al nivel con exposición, manteniendo X fijo
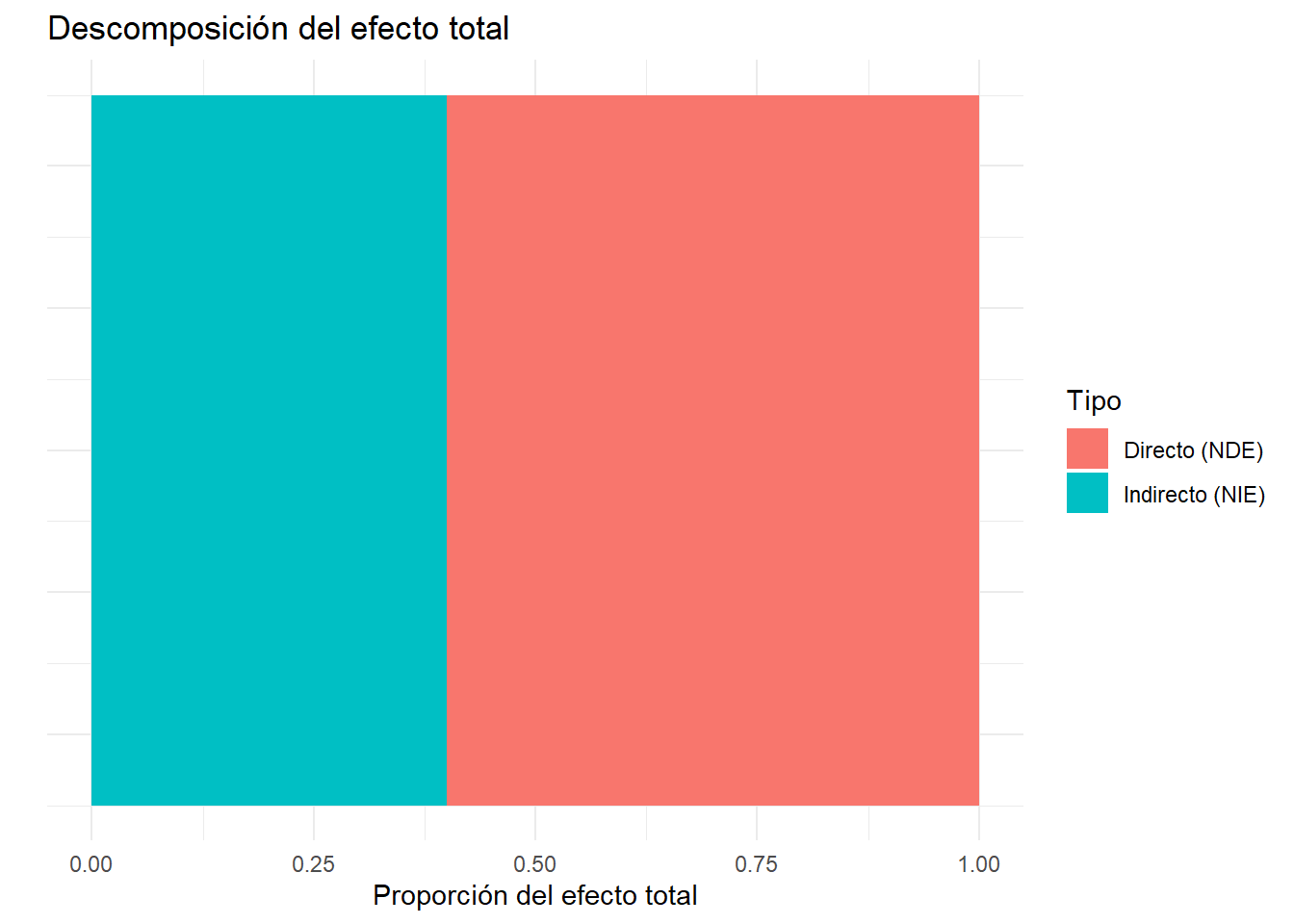
$$\text{Efecto Total} = \text{NDE} + \text{NIE}$$
```{r}
#| label: fig-mediation-decomposition
#| fig-cap: "Descomposición del efecto total"
#| code-fold: true
library(ggplot2)
# Datos para visualización
efectos <- data.frame(
Tipo = c("Directo (NDE)", "Indirecto (NIE)"),
Valor = c(0.6, 0.4),
ymin = 0,
ymax = c(0.6, 1.0)
)
ggplot(efectos, aes(x = 1, y = Valor, fill = Tipo)) +
geom_col(position = "stack", width = 0.5) +
coord_flip() +
labs(y = "Proporción del efecto total",
x = "",
title = "Descomposición del efecto total") +
theme_minimal() +
theme(axis.text.y = element_blank(),
axis.ticks.y = element_blank())
```
## Asunciones para identificación
::: {.callout-important}
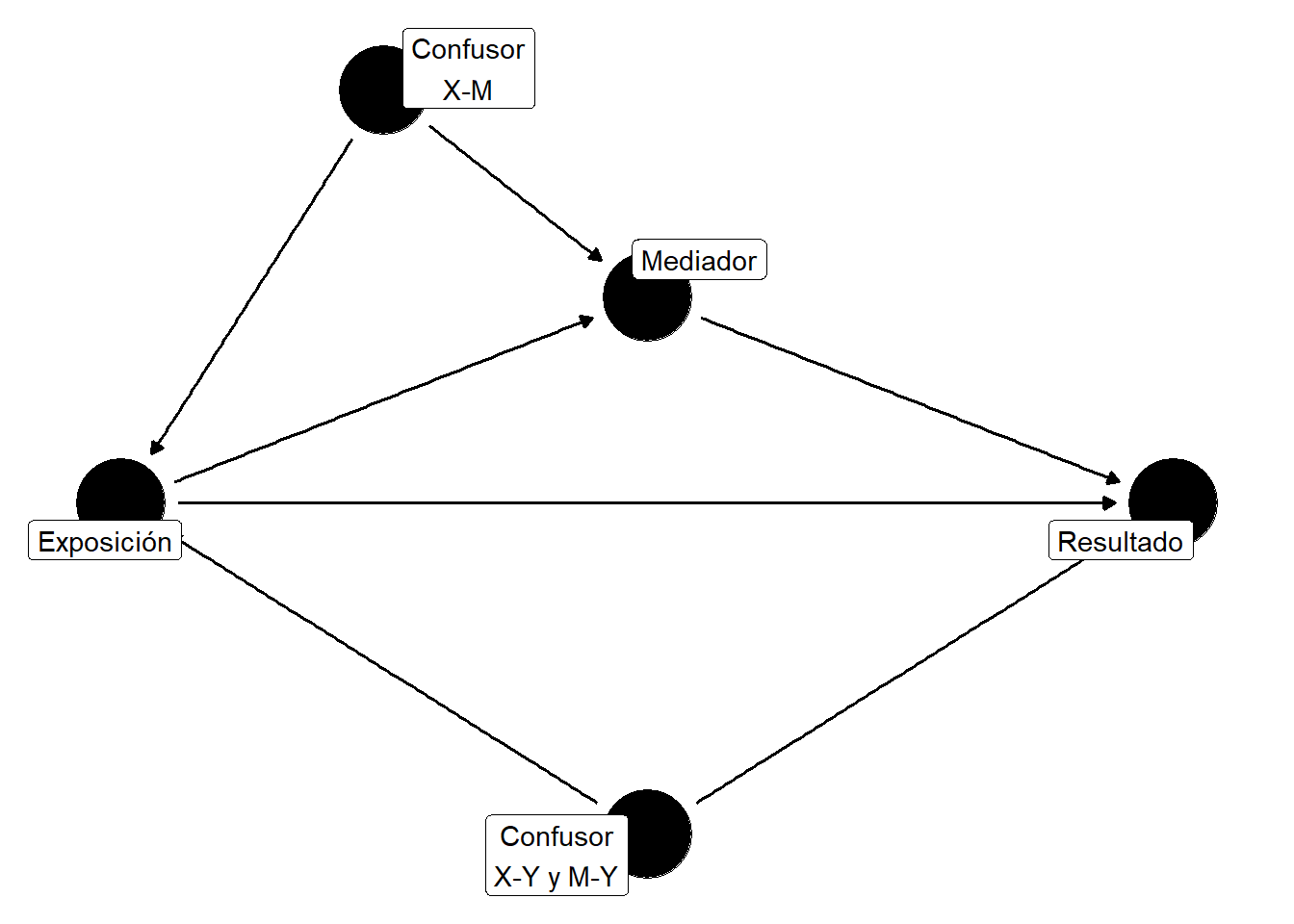
## Cuatro asunciones de no confusión
1. No hay confusión de X → Y
2. No hay confusión de M → Y
3. No hay confusión de X → M
4. No hay confusor de M → Y afectado por X
La cuarta asunción es frecuentemente violada y difícil de verificar.
:::
```{r}
#| label: fig-mediation-assumptions
#| fig-cap: "Escenario con todas las asunciones satisfechas"
#| code-fold: true
full_med_dag <- dagify(
M ~ X + C1,
Y ~ X + M + C2,
X ~ C1 + C2,
coords = list(
x = c(X = 0, M = 1, Y = 2, C1 = 0.5, C2 = 1),
y = c(X = 0, M = 0.5, Y = 0, C1 = 1, C2 = -0.8)
),
labels = c(
X = "Exposición",
M = "Mediador",
Y = "Resultado",
C1 = "Confusor\nX-M",
C2 = "Confusor\nX-Y y M-Y"
)
)
ggdag(full_med_dag, text = FALSE, use_labels = "label") +
theme_dag()
```
## Método de Baron-Kenny (tradicional)
El enfoque clásico usa tres regresiones:
```{r}
#| label: baron-kenny
#| code-fold: false
# Simular datos
set.seed(707)
n <- 1000
X <- rnorm(n) # Exposición
M <- 0.5 * X + rnorm(n, 0, 0.5) # Mediador
Y <- 0.3 * X + 0.6 * M + rnorm(n, 0, 0.5) # Resultado
datos_med <- data.frame(X, M, Y)
# Paso 1: Efecto total (X -> Y)
modelo1 <- lm(Y ~ X, data = datos_med)
cat("Efecto total:", round(coef(modelo1)["X"], 3), "\n")
# Paso 2: Efecto de X sobre M
modelo2 <- lm(M ~ X, data = datos_med)
cat("Efecto X -> M:", round(coef(modelo2)["X"], 3), "\n")
# Paso 3: Efecto de X y M sobre Y
modelo3 <- lm(Y ~ X + M, data = datos_med)
cat("Efecto directo (X -> Y|M):", round(coef(modelo3)["X"], 3), "\n")
cat("Efecto M -> Y:", round(coef(modelo3)["M"], 3), "\n")
# Efecto indirecto (producto de coeficientes)
efecto_indirecto <- coef(modelo2)["X"] * coef(modelo3)["M"]
cat("\nEfecto indirecto (a × b):", round(efecto_indirecto, 3), "\n")
```
### Limitaciones del método tradicional
1. No tiene interpretación causal clara
2. Asume linearidad
3. No funciona bien con mediadores o resultados binarios
4. Intervalos de confianza problemáticos
## Método de mediación causal
El paquete `mediation` implementa el enfoque de efectos naturales:
```{r}
#| label: mediation-package
#| code-fold: false
library(mediation)
# Modelo para el mediador
modelo_mediador <- lm(M ~ X, data = datos_med)
# Modelo para el resultado
modelo_resultado <- lm(Y ~ X + M, data = datos_med)
# Análisis de mediación
med_out <- mediate(modelo_mediador, modelo_resultado,
treat = "X",
mediator = "M",
boot = TRUE,
sims = 500)
summary(med_out)
```
### Interpretación de resultados
```{r}
#| label: mediation-interpretation
#| code-fold: false
cat("=== INTERPRETACIÓN ===\n\n")
cat("ACME (Average Causal Mediation Effect):",
round(med_out$d0, 3), "\n")
cat(" → Efecto que pasa a través del mediador\n\n")
cat("ADE (Average Direct Effect):",
round(med_out$z0, 3), "\n")
cat(" → Efecto que NO pasa por el mediador\n\n")
cat("Proporción mediada:",
round(med_out$n0, 2), "\n")
cat(" → ", round(med_out$n0 * 100), "% del efecto total es mediado\n", sep = "")
```
## Mediación con resultados binarios
```{r}
#| label: binary-mediation
#| code-fold: false
# Datos con resultado binario
set.seed(808)
n <- 2000
X <- rbinom(n, 1, 0.5) # Exposición binaria
M <- plogis(-1 + 1.5 * X) + rnorm(n, 0, 0.2) # Mediador continuo
M <- pmax(0, pmin(1, M)) # Limitar entre 0 y 1
Y <- rbinom(n, 1, plogis(-1.5 + 0.5 * X + 2 * M)) # Resultado binario
datos_bin <- data.frame(X, M, Y)
# Modelos
mod_med_bin <- lm(M ~ X, data = datos_bin)
mod_out_bin <- glm(Y ~ X + M, data = datos_bin, family = binomial)
# Mediación
med_bin <- mediate(mod_med_bin, mod_out_bin,
treat = "X",
mediator = "M",
boot = TRUE,
sims = 200)
summary(med_bin)
```
## Análisis de sensibilidad para mediación
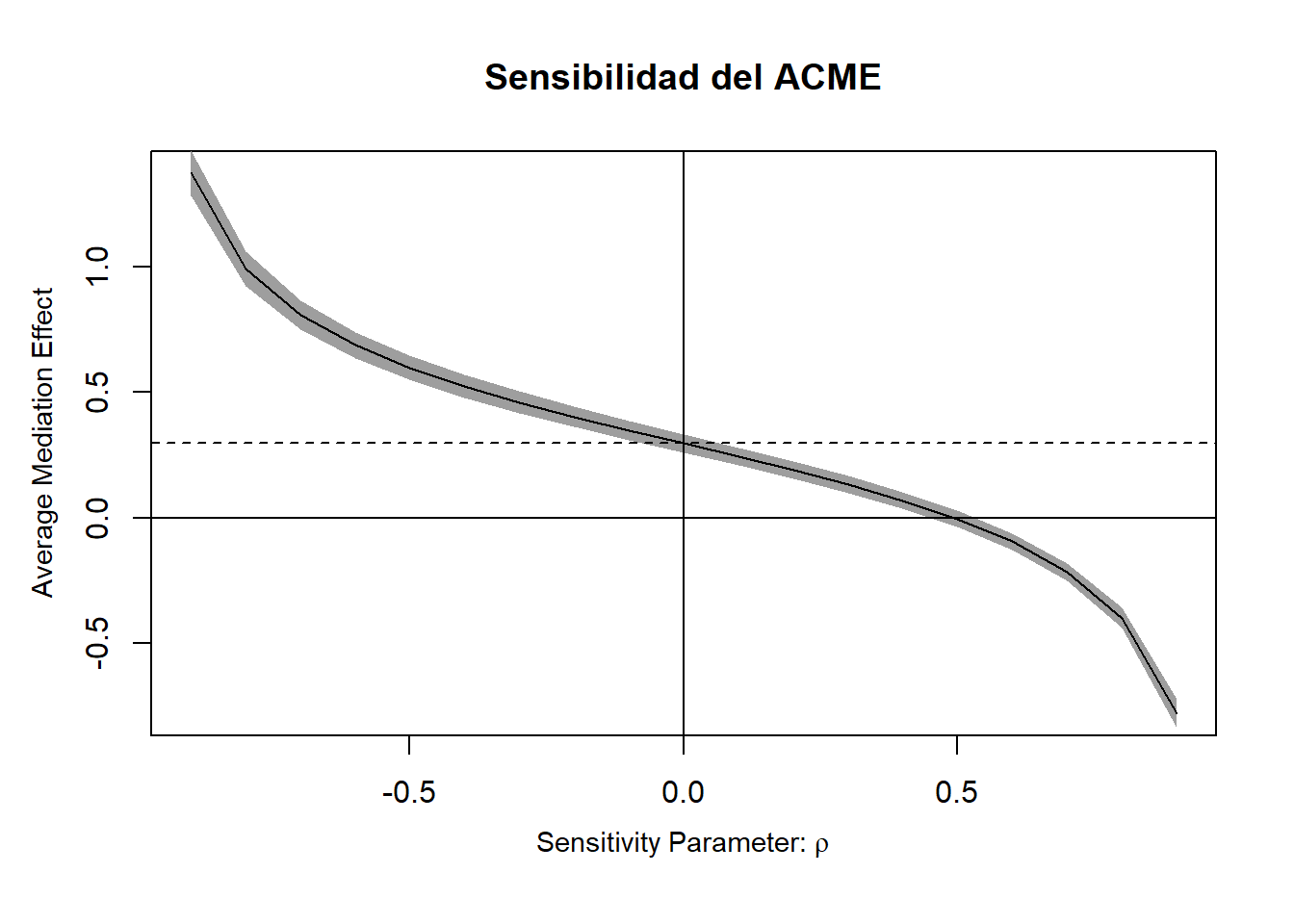
La violación de las asunciones (especialmente la 4ª) puede sesgar las estimaciones. El paquete `mediation` incluye análisis de sensibilidad:
```{r}
#| label: mediation-sensitivity
#| code-fold: false
# Análisis de sensibilidad
sens_med <- medsens(med_out, rho.by = 0.1, sims = 200)
summary(sens_med)
```
```{r}
#| label: fig-mediation-sens
#| fig-cap: "Sensibilidad del efecto indirecto"
#| code-fold: true
plot(sens_med, main = "Sensibilidad del ACME")
```
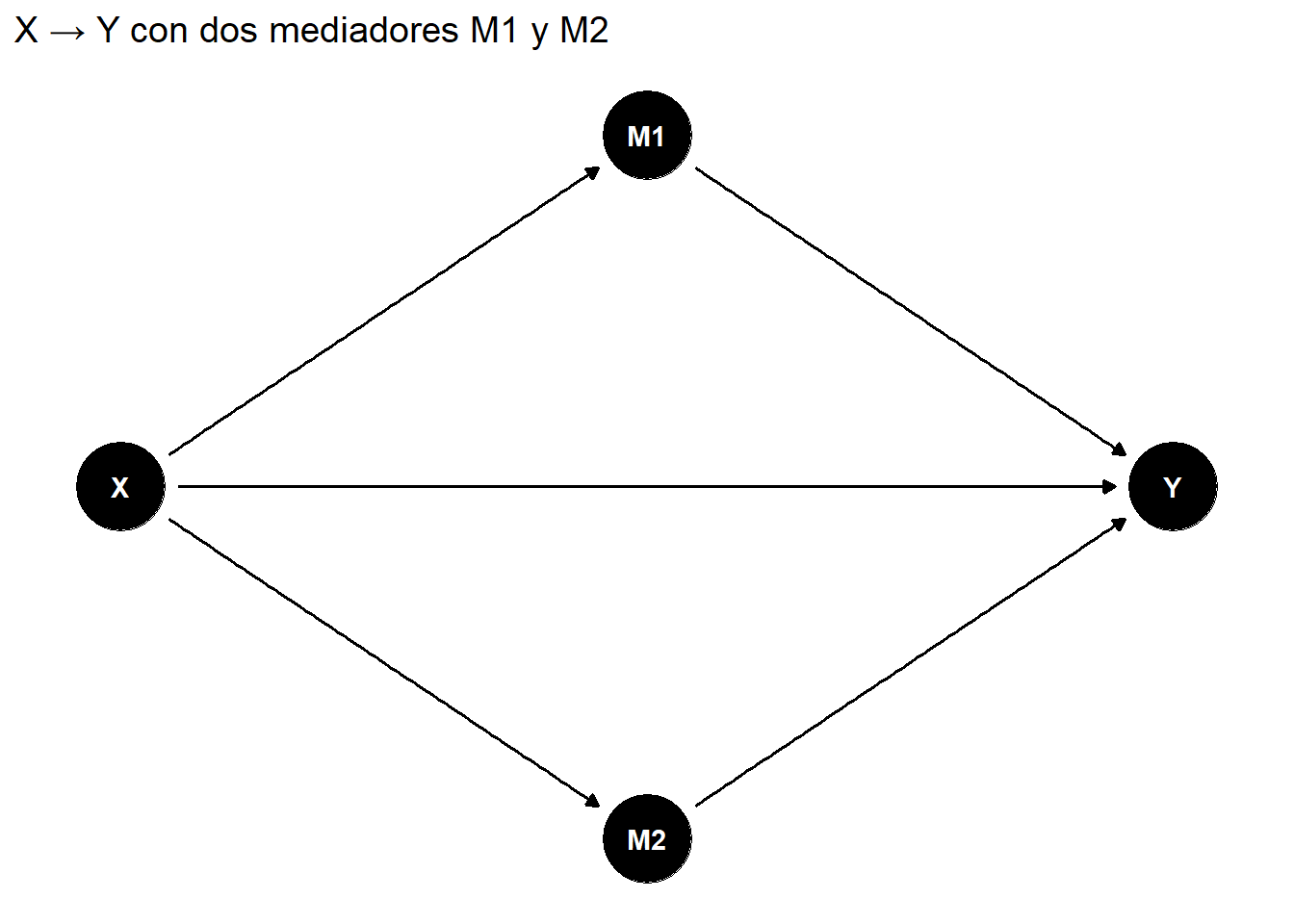
## Múltiples mediadores
Cuando hay varios mediadores, el análisis se complica:
```{r}
#| label: fig-multiple-mediators
#| fig-cap: "Múltiples mediadores"
#| code-fold: true
multi_med_dag <- dagify(
M1 ~ X,
M2 ~ X,
Y ~ X + M1 + M2,
coords = list(
x = c(X = 0, M1 = 1, M2 = 1, Y = 2),
y = c(X = 0, M1 = 0.5, M2 = -0.5, Y = 0)
)
)
ggdag(multi_med_dag) +
theme_dag() +
labs(title = "X → Y con dos mediadores M1 y M2")
```
::: {.callout-warning}
## Complicación
Con múltiples mediadores que pueden afectarse entre sí, los efectos indirectos específicos para cada mediador generalmente no están identificados sin asunciones adicionales.
:::
## Ejercicios
::: {.callout-tip}
## Ejercicio 1
Un programa educativo (X) mejora las calificaciones (Y). Se hipotetiza que el efecto es mediado por horas de estudio (M).
1. Simula datos bajo este escenario
2. Estima NDE y NIE
3. Calcula la proporción mediada
4. Realiza análisis de sensibilidad
:::
::: {.callout-tip}
## Ejercicio 2
Considera el caso donde el mediador M tiene un confusor U que también está afectado por X (violación de la 4ª asunción). Simula este escenario y muestra cómo sesga las estimaciones.
:::
## Resumen
- La mediación descompone el efecto total en directo e indirecto
- NDE y NIE tienen interpretación causal clara
- Se requieren cuatro asunciones de no confusión
- La 4ª asunción (no confusor M→Y afectado por X) es crítica
- El paquete `mediation` implementa estos métodos
- Siempre realizar análisis de sensibilidad
## Referencias {.unnumbered}